细菌通过水平基因转移(horizontal gene transfer, HGT)获取新的DNA,如果转移的DNA被保留在受体细胞中,则受体细胞的生理和活性可能会被转移的DNA中编码的基因显著改变(1,2)。这一过程有助于细菌的基因组多样性,使其快速适应不断变化的环境条件并提供竞争优势,同时也促进了细菌病原体中致病性基因岛和抗生素抗性基因的传播(3,4),由此引起的抗菌素耐药性导致的公共卫生挑战被广泛关注。因此,了解HGT机制有助于解释细菌进化,对于控制病原体和抗生素耐药性也至关重要。
细菌的HGT途径主要为由质粒/转座子介导的接合,噬菌体介导的转导和转化,而转化需要受体菌建立感受态使其能够吸收胞外DNA(5)。该课题组之前的研究发现了在枯草芽胞杆菌(Bacillus subtilis)中一种高频的细胞间基因转移的现象,有趣的是,这个过程要求受体菌建立感受态,但是几乎完全抵抗DNase的处理,并且似乎需要在供体和受体细胞之间紧密接近(close proximity),这种供体细胞和受体细胞之间存在协调过程的现象被称为细胞间自然转化(cell-to-cell natural transformation, CTCNT)(6)。CTCNT采用活的供体细胞充当转化体系中的DNA来源,其转化频率相较于枯草芽胞杆菌的二步法转化或者外源游离DNA做为转化DNA来源的自然转化(DNA-to-cell natural transformation, DTCNT),在单个或者多个筛选标记下都具有明显优势,但是,目前尚不清楚CTCNT高转化频率背后的机制。
2023年3月13日,武汉大学生命科学学院、病毒学国家重点实验室陈向东教授和杜世燊研究员在Nucleic Acids Research 上在线发表了题为“Cell-to-cell natural transformation in Bacillus subtilis facilitates large scale of genomic exchanges and the transfer of long continuous DNA regions”的研究论文,该研究报道了在枯草芽胞杆菌CTCNT通过增加转移事件的数量和促进大型连续基因组区域的转移来促进枯草芽胞杆菌的HGT,并指出了活细胞作为转化DNA的来源在自然环境中的潜力。

在该课题组之前的研究中观察到枯草芽胞杆菌细胞间遗传转化中,遗传距离相距130kb的三个筛选标记可以通过一次CTCNT-C(CTCNT with chromosome)实验共同转移到一个受体细胞中。在本研究中,进一步量化比较了用游离DNA和活细胞作为DNA来源的转化效率,在单个筛选标记下,CTCNT-C的转化效率至少比使用饱和浓度的纯化染色体DNA(DTCNT-C)的转化效率高两个数量级,而在两个或者三个筛选标记下DTCNT-C的转化频率大大降低,表明CTCNT-C促进大染色体DNA片段的转移。为了进一步测试CTCNT-C在超过遗传距离超过200kb的筛选标记是否可以同时转移,作者将筛选标记的遗传遗传距离进一步加大,使其在62kb到近2000kb 之间。在现在两个筛选标记的情况下,无论标记间的遗传距离如何,都能在选择平板上得到大量的转化子,而随着选择压力的增加,转化子也逐渐减少(图1A)。这些结果表明,CTCNT-C在HGT中非常高效,并促进多个远距离遗传标记的共遗传。
由于在上述遗传实验中同时选择了多个筛选标记,因此共遗传效率可能存在偏差。为此作者测试了未选择的标记是否会通过CTCNT-C与trpC选择标记共同转移。与图1A中的直接同时选择的结果相一致,平板上的菌落数量随着选择压力的增加而减少,而那些被超过475kb隔开的遗传标记很少能通过单次重组事件进行整体转化(图1C)。
为了进一步研究共遗传率与选择和未选择标记之间的在不同遗传距离的关系,研究人利用CTCNT-C测试了营养型标记与整合到枯草芽胞杆菌染色体中的外源性非选择性标记(红/绿荧光蛋白)的共遗传。未选择标记与选定标记之间的距离在500kb范围内时,共同遗传比率随着距离增加而显着下降,当距离大于500kb时,共同遗传比率在非常低的频率上保持不变。
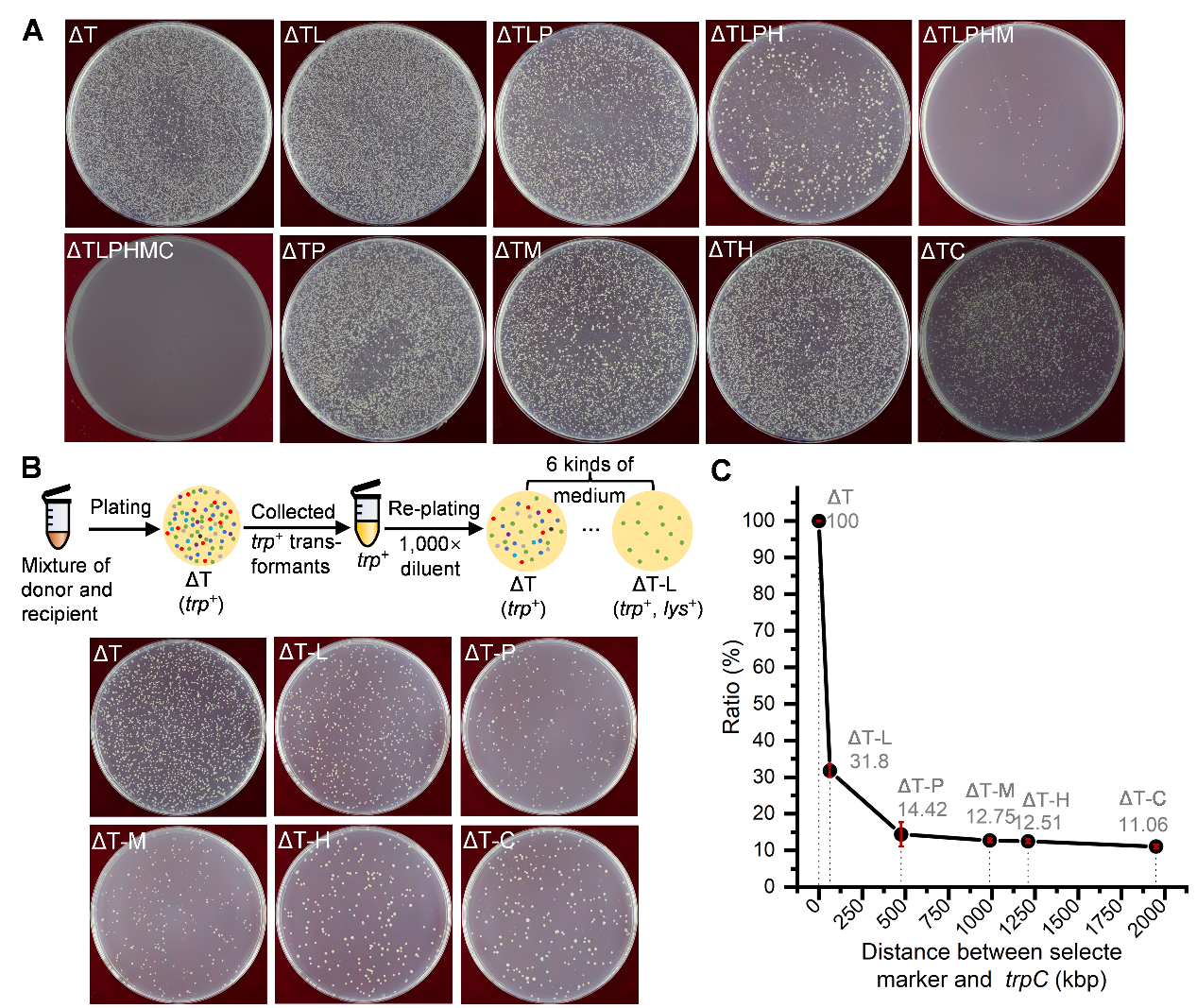
图1 枯草芽胞杆菌CTCNT-C多个遗传标记的共转化
(图源:Liping Deng et al., Nucleic Acids Research, 2023)
为了确定CTCNT-C高效率的潜在机制,使用全基因组测序(whole genome sequencing,WGS)来确定CTCNT-C获得的转化子中转移的DNA片段的长度和重组事件的数量。供体菌和受体菌的基因组上均匀分布了70909个单核苷酸多态性(single nucleotide polymorphism,SNP),这些SNP提供了遗传标记的高密度图谱,平均密度约为每56bp中含有1个,最终通过比较转化子中连续的SNP来确定供体特异性片段。
当只有trpC作为单个筛选标记时,通过CTCNT-C得到的转化子中trpC侧翼的连续DNA片段最长达246.4kb,而DTCNT-C的只有17.01kb(图2)。这些结果证实了CTCNT-C促进连续的大DNA片段的转移,与前面的遗传学实验的结果相一致。除了选择的trpC位点外,CTCNT-C还促进了大量未选择的DNA片段的转移,每个CTCNT-C转化子都额外还有4-30次的转化事件,而DTCNT-C转化子绝大多数小于5次。这些数据表明,枯草芽胞杆菌CTCNT-C过程的重组菌株中,重组事件非常频繁的发生在所选标记以外的位置,这也证实了多次转化是遗传标记共同遗传的主要机制。
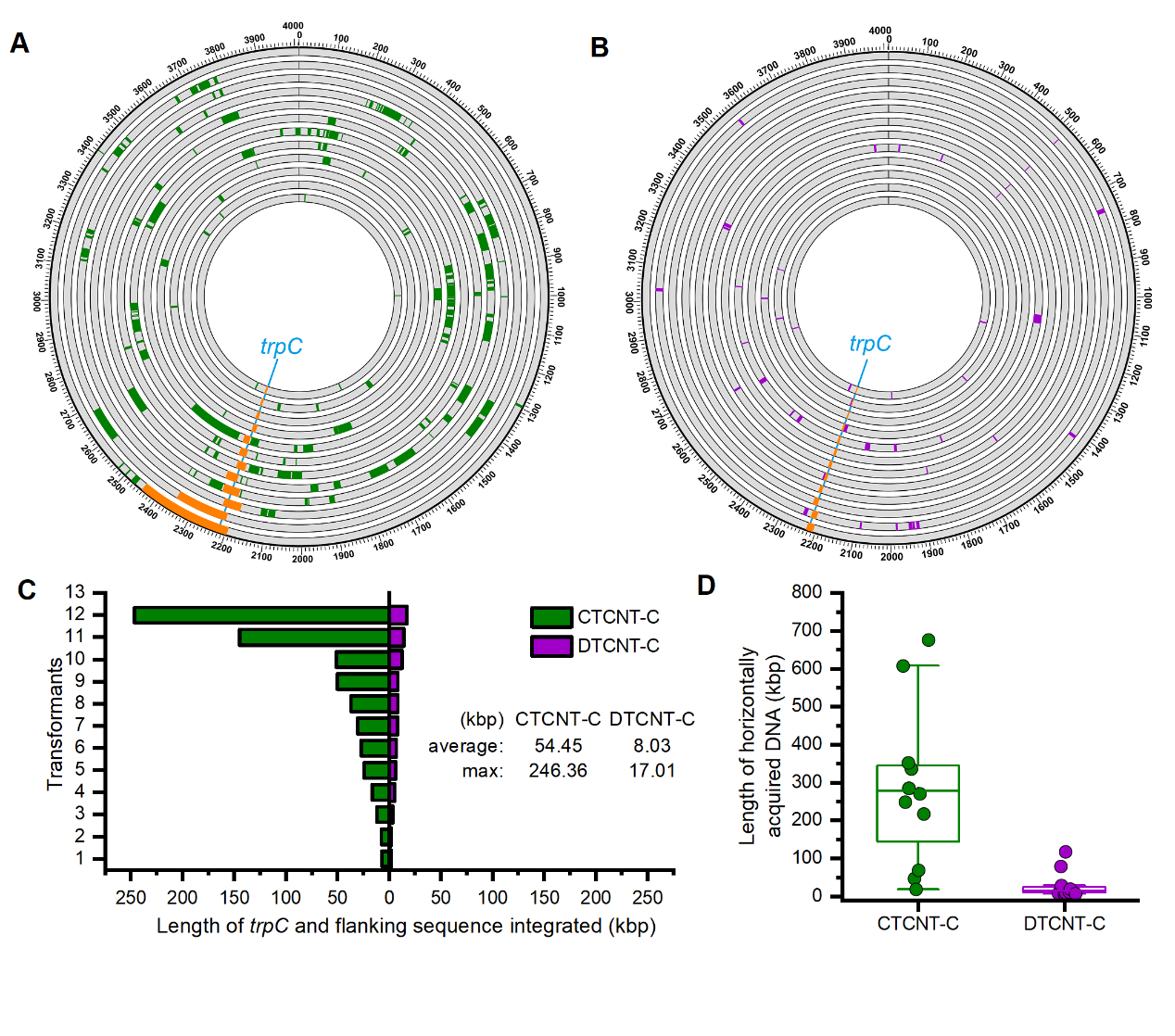
图2 CTCNT-C和DTCNT-C在单个筛选标记下转移的DNA片段
(图源:Liping Deng et al., Nucleic Acids Research, 2023)
在12个CTCNT-C的转化子中测序数据中可以看出供体特异性SNPs遍布在基因组(图2A),假设转化片段DNA重叠部分仅计算一次,则转移区域的总长度达到2.07Mb,占供体基因组的51.58%。这表明大约一半的供体基因组可以被转化。为了探究枯草芽胞杆菌CTCNT-C中HGT事件的最大数量和获得的基因组区域的最大长度,对于同时选择多个标记物的重组子进行WGS分析。与单个筛选标记结果类似,转化子中除了被选中的多个筛选区域被回补外,在所选标记物以外的染色体位置的重组事件普遍存在(图3)。每个重组体平均发生了40次转化事件,最多的进行了66次整合,最大转移片段高达346.93kb。每个转化子获得的供体DNA的总长度显著增加,从0.7到1.5Mb不等,其中一个重组菌的基因组上36.52%的区域(1539.4kb)被来自供体菌株的同源序列所取代。相比之下,平均只有280kb的供体DNA片段镶嵌在单个trpC筛选下的重组菌株染色体上。另外,连续的长DNA片段附近有许多小片段,有些可能是当初被细胞质切割成小片段的,如果将这些小片段聚类成重组束,平均转化片段长度则增加了1.3倍。综上所述,与DTCNT-C相比,CTCNT-C促进了更大规模的基因组替换,特别是当同时选择多个标记时。
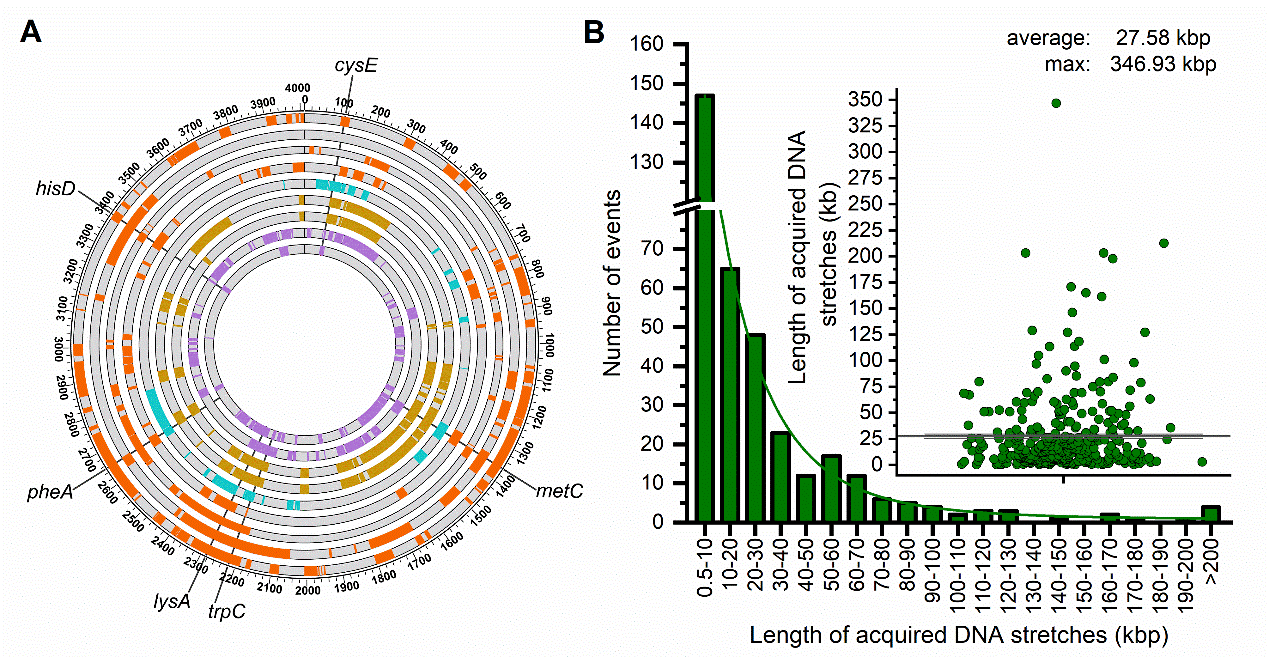
图3 多个筛选标记下CTCNT-C转移的DNA片段
(图源:Liping Deng et al., Nucleic Acids Research, 2023)
结论与展望
该研究揭示了枯草芽胞杆菌CTCNT相较于DTCNT转化更高效的原因,基因组上遗传距离上相距甚远的基因的共同转化是由于转移和重组包含该标记的大的连续DNA片段并伴随着大量未选择区域的交换。这些结果表明自然转化可能在促进枯草芽胞杆菌的进化和适应中发挥重要作用。枯草芽胞杆菌细胞间遗传转化的潜在机制有待进一步确定,同时,利用健康的活细胞作为转化DNA的来源进行其他天然感受态细菌的自然转化,有助于重新评估自然转化在HGT中的重要性。
武汉大学生命科学学院陈向东教授和杜世燊研究员为该论文共同通讯作者,博士生邓丽萍为第一作者。这项工作得到了国家自然科学基金面上项目(31970030)的支持。
原文链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad138/7076482?searchresult=1
1. Arnold, B.J., Huang, I.T. and Hanage, W.P. (2022) Horizontal gene transfer and adaptive evolution in bacteria. Nat. Rev. Microbiol., 20, 206-218.
http://www.ncbi.nlm.nih.gov/pubmed/34773098
http://dx.doi.org/10.1038/s41579-021-00650-4
2. Baltrus, D.A. (2013) Exploring the costs of horizontal gene transfer. Trends Ecol. Evol., 28, 489-495.
http://www.ncbi.nlm.nih.gov/pubmed/23706556
http://dx.doi.org/10.1016/j.tree.2013.04.002
3. von Wintersdorff, C.J.H., Penders, J., van Niekerk, J.M., Mills, N.D., Majumder, S., van Alphen, L.B., Savelkoul, P.H.M. and Wolffs, P.F.G. (2016) Dissemination of antimicrobial resistance in microbial ecosystems through horizontal gene transfer. Frontiers in microbiology, 7, 173.
http://www.ncbi.nlm.nih.gov/pubmed/26925045
http://dx.doi.org/10.3389/fmicb.2016.00173
4. Chun, J., Grim, C.J., Hasan, N.A., Lee, J.H., Choi, S.Y., Haley, B.J., Taviani, E., Jeon, Y.-S., Kim, D.W., Lee, J.-H. et al. (2009) Comparative genomics reveals mechanism for short-term and long-term clonal transitions in pandemic Vibrio cholerae. Proc. Natl. Acad. Sci. U. S. A., 106, 15442-15447.
http://www.ncbi.nlm.nih.gov/pubmed/19720995
http://dx.doi.org/10.1073/pnas.0907787106
5. Cabezón, E., Ripoll-Rozada, J., Peña, A., de la Cruz, F. and Arechaga, I. (2015) Towards an integrated model of bacterial conjugation. FEMS Microbiol. Rev., 39, 81-95.
http://www.ncbi.nlm.nih.gov/pubmed/25154632
http://dx.doi.org/10.1111/1574-6976.12085
6. Zhang, X., Jin, T., Deng, L., Wang, C., Zhang, Y. and Chen, X. (2018) Stress-induced, highly efficient, donor cell-dependent cell-to-cell natural transformation in Bacillus subtilis. J. Bacteriol., 200.
http://www.ncbi.nlm.nih.gov/pubmed/29941421
http://dx.doi.org/10.1128/JB.00267-18