2019年10月30日,国际知名期刊Nucleic Acids Research(核酸研究,影响因子11.147)在线发表了来自武汉大学生命科学学院周宇教授和朱玉贤院士课题组在锦葵科植物功能基因组学数据库建设方面联合取得的最新研究成果。该工作为植物学家开展锦葵科植物比较基因组学分析提供了有力的在线分析工具。
论文题为“MaGenDB: a functional genomics hub for Malvaceae plants(MaGenDB:锦葵科植物功能基因组学集成数据库)”。生命科学学院博士研究生王得和、樊伟良和郭晓龙为论文共同第一作者,周宇教授、朱玉贤院士和王坤副研究员为论文的共同通讯作者。该工作得到了国家自然科学基金、科技部、武汉大学自主科研交叉学科等项目的资助。
锦葵科是植物界重要的一个分支类群,包括许多重要的经济作物,如种子和茎皮纤维来源的棉花和黄麻、制造可可粉和“巧克力糖”原料的可可、热带著名“水果之王”榴莲、观赏花卉植物木槿和优良木材来源的椴树等。随着全球越来越多锦葵科植物基因组的陆续解析,及相关转录调控等高通量数据的积累,研究者迫切需要一个能够整合基因组、表观修饰组、转录组和蛋白质组等多层次数据的比较工具,对植物特异科属的特定生物学过程或基因家族的基因序列、结构、转录调控等进行比较分析。为此,周宇教授和朱玉贤院士、王坤副研究员合作,共同构建了首个锦葵科植物功能基因组学网站-MaGenDB (http://magen.whu.edu.cn)。截至目前,MaGenDB共收录了锦葵科9个属共13个物种的基因组以及相应的全基因组关联分析位点(GWAS)和单核苷酸变异位点(SNP)信息;收录并处理了包括RNA-seq和蛋白质谱等在内的9种共374套组学数据;预测鉴定出2485万个不同类型的功能元件、2897万个基因共线性关系以及1.7亿个蛋白-蛋白相互作用对。该数据库独立设计并开发出多种可视化组件与基因组在线比较工具,并通过有效关联将不同类型的数据有机整合到了一起,为用户提供了友好的数据检索、下载和在线分析服务。
该工作也是两个研究团队,继不久前刚刚在自然子刊《Nature Communications》合作发表有关亚洲棉的精细转录组图谱工作之后,所取得的又一个重要研究成果。
论文在线网址:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkz953/5609523
图片介绍如下:
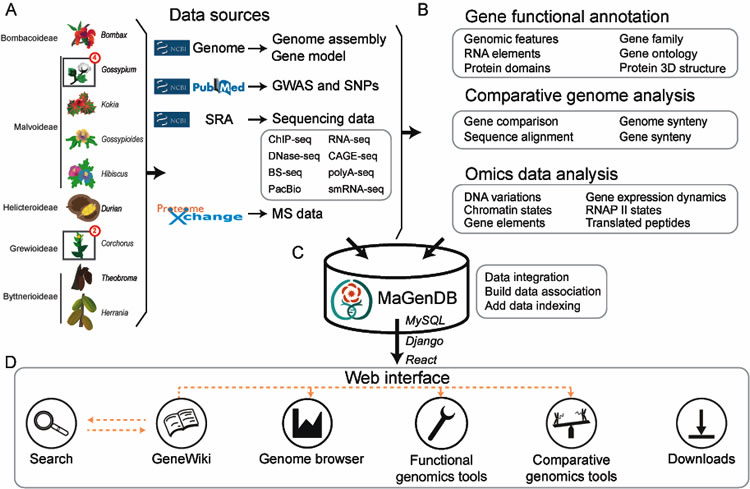
图注:MaGenDB的构建和功能介绍